Fuente: Agencia Dicyt
Investigadores del Instituto de Biología y Genética Molecular (IBGM), centro mixto de la Universidad de Valladolid (UVa) y el CSIC, de la Universidad Complutense de Madrid (UCM) y de la Universidad de Sevilla, han encontrado una región específica del gen ApoD que se sobreexpresa en el cerebelo en condiciones de estrés oxidativo y que estaría realizando una función neuroprotectora, lo que podría constituir, en el futuro, la base sobre la que diseñar nuevos tratamientos frente a diversas enfermedades neurodegenerativas. El trabajo se ha publicado en la revista científica ‘PLOS ONe’.
Las lipocalinas son una gran familia de proteínas presente en los animales vertebrados, compuesta por más de 150 miembros. Pero de este gran número de lipocalinas, algunas de ellas de gran interés en el ámbito biomédico, solo unas pocas se expresan dentro del sistema nervioso. Una de ellas es la apolipoproteína D, que está codificada por el gen ApoD -es decir, el gen ApoD es el que contiene la información precisa para producir esta proteína y las “instrucciones” acerca de su función-. Pero, ¿cuál es esa función?
La comunidad científica sabe bien que ApoD es uno de los pocos genes que se sobreexpresa constantemente en el cerebro envejecido de todas las especies de vertebrados en los que se ha analizado, así como en la mayoría de enfermedades neurodegenerativas y psiquiátricas, independientemente de su origen.
Esquizofrenia, trastorno bipolar, enfermedad de Alzheimer o esclerosis múltiple comparten esa sobreexpresión de ApoD. Pero su función aún no está del todo clara, ¿está causando de alguna forma ApoD estas enfermedades? ¿o ApoD está tratando de realizar una función protectora precisamente frente a ellas?
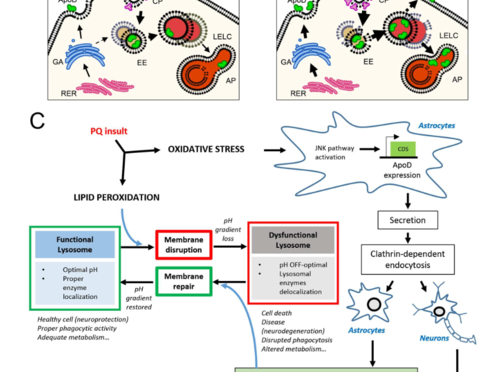
Sergio Díez Hermano, primer autor de este estudio, detalla a DiCYT que el estrés oxidativo aparece claramente como un factor común en el envejecimiento cerebral y en estas enfermedades de etiologías tan variadas. Y el ratón, como modelo animal, ha aportado una gran cantidad de conocimientos sobre las funciones de esta lipocalina atípica. Según los datos recogidos en trabajos previos, ApoD retrasaría el envejecimiento cerebral y promovería la longevidad y la supervivencia de las neuronas, protegiéndolas en condiciones de estrés oxidativo.
Así, controlar la expresión de ApoD podría llevar, en el futuro, a nuevos tratamientos para estas enfermedades. Pero para controlar la expresión de ApoD es necesario, primero, obtener más información sobre cómo se regulan y coordinan todas estas funciones.
Por ello, el equipo realizó un extenso trabajo ‘in silico’ –o lo que es lo mismo, mediante análisis bioinformático-, acompañado de comprobaciones experimentales, para analizar regiones inexploradas reguladoras del gen ApoD en ratón, en diferentes tejidos, etapas de desarrollo y condiciones fisiológicas
“Hemos encontrado que hay una región concreta del gen ApoD que se activa en condiciones de estrés oxidativo. Está más expresada en el cerebelo y solo cuando hay estrés oxidativo, lo que es una prueba indirecta del papel protector de ApoD”, explica Díez Hermano. Esta región, denominada variante E, tendría un papel potencial como regulador de una respuesta rápida y protectora de ApoD frente al envejecimiento y a muchas enfermedades neurodegenerativas, causalmente relacionadas con el estrés oxidativo.
Los hallazgos de este trabajo abren nuevas y prometedoras líneas de investigación, por ejemplo, de terapias farmacológicas que permitan expresar ApoD anticipándose a su expresión endógena. El equipo también avanza en otras líneas en relación a este gen, investigando por ejemplo la interrelación entre diabetes, inflamación y ApoD e incluso tratando de desentrañar la historia evolutiva de las lipocalinas.
Referencia: Diez-Hermano S, Mejias A, Sanchez D, Gutierrez G, Ganfornina MD (2020) Control of the neuroprotective Lipocalin Apolipoprotein D expression by alternative promoter regions and differentially expressed mRNA 5’ UTR variants. PLoS ONE 15(6): e0234857. https://doi.org/10.1371/journal.pone.0234857